ベイズ線形回帰#
import matplotlib.pyplot as plt
import numpy as np
import scipy as sp
# データを作成
n = 1000
from scipy.stats import multivariate_normal
mean = np.array([3, 5])
Sigma = np.array([
[1, 0.5],
[0.5, 2],
])
X = multivariate_normal.rvs(mean=mean, cov=Sigma, size=n, random_state=0)
import statsmodels.api as sm
X = sm.add_constant(X)
# 真のパラメータ
beta = np.array([2, 3, 4])
データが均一分散の場合#
# 均一分散の場合
e = np.random.normal(loc=0, scale=1, size=n)
y = X @ beta + e
# 頻度主義
import statsmodels.api as sm
ols = sm.OLS(y, X).fit(cov_type="HC1")
ols.summary()
| Dep. Variable: | y | R-squared: | 0.982 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.982 |
| Method: | Least Squares | F-statistic: | 2.814e+04 |
| Date: | Wed, 18 Feb 2026 | Prob (F-statistic): | 0.00 |
| Time: | 14:43:33 | Log-Likelihood: | -1383.6 |
| No. Observations: | 1000 | AIC: | 2773. |
| Df Residuals: | 997 | BIC: | 2788. |
| Df Model: | 2 | ||
| Covariance Type: | HC1 |
| coef | std err | z | P>|z| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| const | 1.9161 | 0.125 | 15.323 | 0.000 | 1.671 | 2.161 |
| x1 | 3.0252 | 0.033 | 92.438 | 0.000 | 2.961 | 3.089 |
| x2 | 4.0005 | 0.023 | 173.814 | 0.000 | 3.955 | 4.046 |
| Omnibus: | 3.352 | Durbin-Watson: | 1.970 |
|---|---|---|---|
| Prob(Omnibus): | 0.187 | Jarque-Bera (JB): | 3.304 |
| Skew: | -0.108 | Prob(JB): | 0.192 |
| Kurtosis: | 2.820 | Cond. No. | 25.9 |
Notes:
[1] Standard Errors are heteroscedasticity robust (HC1)
import pymc as pm
import arviz as az
model = pm.Model()
with model:
beta0 = pm.Normal("beta0", mu=0, sigma=1)
beta1 = pm.Normal("beta1", mu=0, sigma=1)
beta2 = pm.Normal("beta2", mu=0, sigma=1)
sigma = pm.HalfNormal("sigma", sigma=1) # 分散なので非負の分布を使う
# 平均値 mu
mu = beta0 + beta1 * X[:, 1] + beta2 * X[:, 2]
# 観測値をもつ確率変数は_obsとする
y_obs = pm.Normal("y_obs", mu=mu, sigma=sigma, observed=y)
# モデルをGraphvizで表示
pm.model_to_graphviz(model)
# ベイズ線形回帰モデルをサンプリング
with model:
idata = pm.sample(
chains=2,
tune=1000, # バーンイン期間の、捨てるサンプル数
draws=2000, # 採用するサンプル数
random_seed=0,
)
# 各chainsの結果を表示
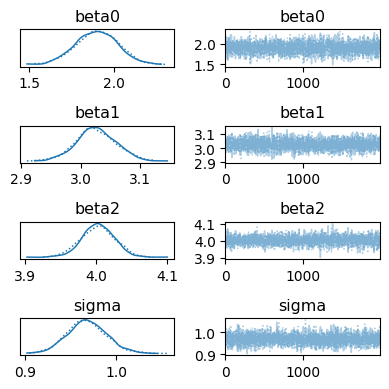
az.plot_trace(idata, figsize=[4, 4])
plt.tight_layout()
plt.show()
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (2 chains in 2 jobs)
NUTS: [beta0, beta1, beta2, sigma]
Sampling 2 chains for 1_000 tune and 2_000 draw iterations (2_000 + 4_000 draws total) took 4 seconds.
We recommend running at least 4 chains for robust computation of convergence diagnostics
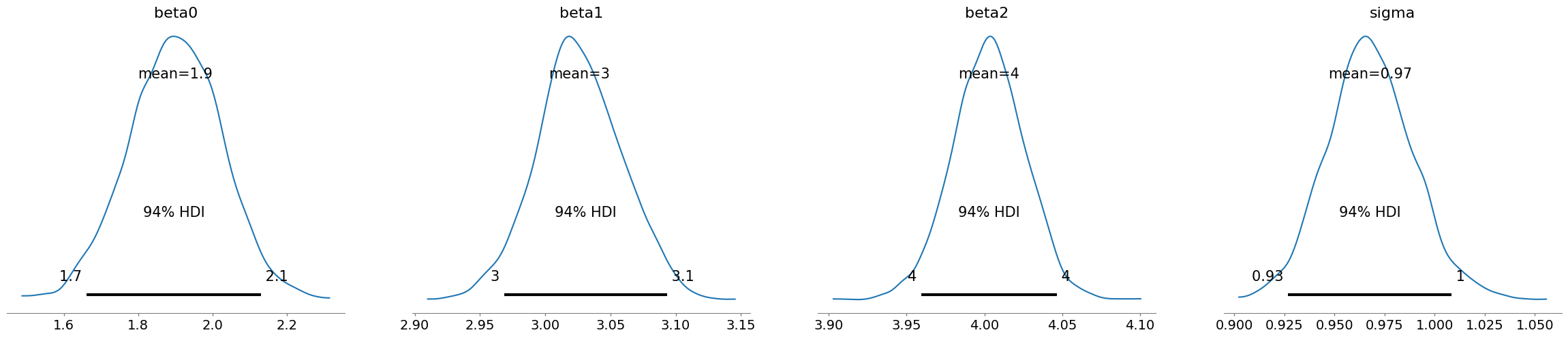
az.plot_posterior(idata)
plt.show()
データが不均一分散の場合#
# 不均一分散の場合
def normalize(x):
return (x - x.min()) / (x.max() - x.min())
sigma = 1 + normalize(X[:, 1] + X[:, 2]) * 3
e = np.random.normal(loc=0, scale=sigma, size=n)
y = X @ beta + e
頻度主義 & 不均一分散に頑健な誤差推定#
# 頻度主義
import statsmodels.api as sm
ols = sm.OLS(y, X).fit(cov_type="HC1")
ols.summary()
| Dep. Variable: | y | R-squared: | 0.883 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.883 |
| Method: | Least Squares | F-statistic: | 3745. |
| Date: | Wed, 18 Feb 2026 | Prob (F-statistic): | 0.00 |
| Time: | 14:43:50 | Log-Likelihood: | -2365.1 |
| No. Observations: | 1000 | AIC: | 4736. |
| Df Residuals: | 997 | BIC: | 4751. |
| Df Model: | 2 | ||
| Covariance Type: | HC1 |
| coef | std err | z | P>|z| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| const | 2.0272 | 0.316 | 6.421 | 0.000 | 1.408 | 2.646 |
| x1 | 2.9575 | 0.088 | 33.623 | 0.000 | 2.785 | 3.130 |
| x2 | 4.0229 | 0.065 | 61.850 | 0.000 | 3.895 | 4.150 |
| Omnibus: | 3.124 | Durbin-Watson: | 2.039 |
|---|---|---|---|
| Prob(Omnibus): | 0.210 | Jarque-Bera (JB): | 3.466 |
| Skew: | 0.006 | Prob(JB): | 0.177 |
| Kurtosis: | 3.288 | Cond. No. | 25.9 |
Notes:
[1] Standard Errors are heteroscedasticity robust (HC1)
↑ 切片の推定にバイアスが入っている
均一分散を想定したベイズ線形回帰#
import pymc as pm
import arviz as az
model = pm.Model()
with model:
beta0 = pm.Normal("beta0", mu=0, sigma=1)
beta1 = pm.Normal("beta1", mu=0, sigma=1)
beta2 = pm.Normal("beta2", mu=0, sigma=1)
sigma = pm.HalfNormal("sigma", sigma=1) # 分散なので非負の分布を使う
# 平均値 mu
mu = beta0 + beta1 * X[:, 1] + beta2 * X[:, 2]
# 観測値をもつ確率変数は_obsとする
y_obs = pm.Normal("y_obs", mu=mu, sigma=sigma, observed=y)
# モデルをGraphvizで表示
pm.model_to_graphviz(model)
# ベイズ線形回帰モデルをサンプリング
with model:
idata = pm.sample(
chains=2,
tune=1000, # バーンイン期間の、捨てるサンプル数
draws=2000, # 採用するサンプル数
random_seed=0,
)
# 各chainsの結果を表示
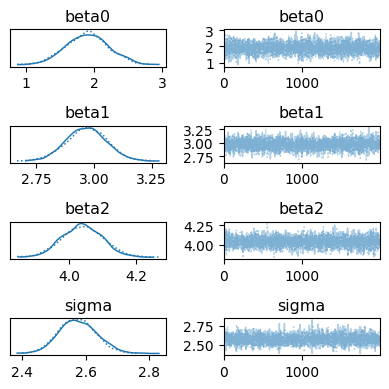
az.plot_trace(idata, figsize=[4, 4])
plt.tight_layout()
plt.show()
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (2 chains in 2 jobs)
NUTS: [beta0, beta1, beta2, sigma]
Sampling 2 chains for 1_000 tune and 2_000 draw iterations (2_000 + 4_000 draws total) took 4 seconds.
We recommend running at least 4 chains for robust computation of convergence diagnostics
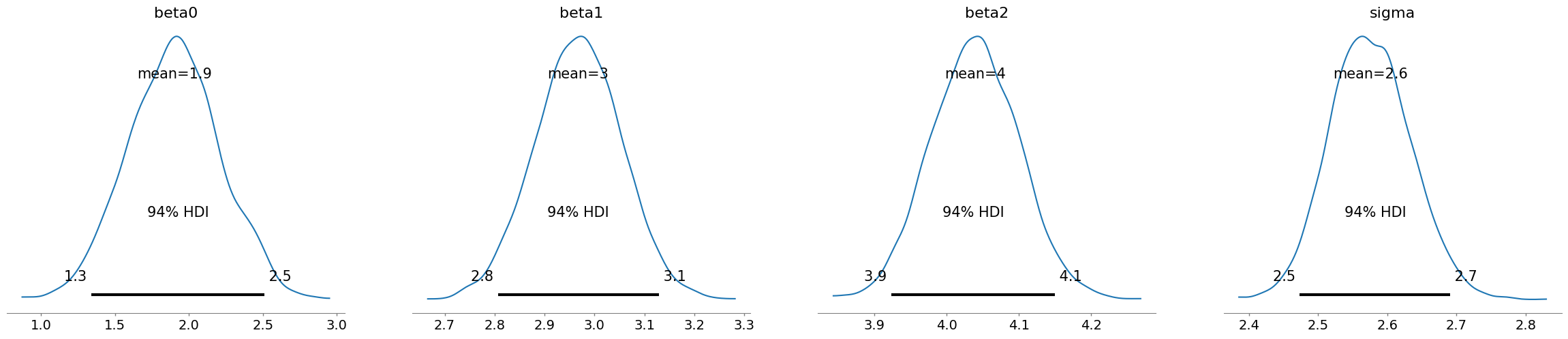
az.plot_posterior(idata)
plt.show()
不均一分散を想定したベイズ線形回帰(WIP)#
分散をxの関数にしたかった。以下コードで推定できるが個々の\(\sigma_i\)が別々に推定される形になって結果が見づらい。もっといい表し方はないものか。
import pymc as pm
import arviz as az
model = pm.Model()
with model:
beta0 = pm.Normal("beta0", mu=0, sigma=1)
beta1 = pm.Normal("beta1", mu=0, sigma=1)
beta2 = pm.Normal("beta2", mu=0, sigma=1)
# 誤差分散にも線形モデルを入れる
w0 = pm.Normal("w0", mu=0, sigma=1)
w1 = pm.Normal("w1", mu=0, sigma=1)
w2 = pm.Normal("w2", mu=0, sigma=1)
lam = pm.math.exp(w0 + w1 * X[:, 1] + w2 * X[:, 2])
sigma = pm.Exponential("sigma", lam=lam) # 分散なので非負の分布を使う
# 平均値 mu
mu = beta0 + beta1 * X[:, 1] + beta2 * X[:, 2]
# 観測値をもつ確率変数は_obsとする
y_obs = pm.Normal("y_obs", mu=mu, sigma=sigma, observed=y)
# モデルをGraphvizで表示
pm.model_to_graphviz(model)
# ベイズ線形回帰モデルをサンプリング
with model:
idata = pm.sample(
chains=2,
tune=1000, # バーンイン期間の、捨てるサンプル数
draws=2000, # 採用するサンプル数
random_seed=0,
)
# 各chainsの結果を表示
az.plot_trace(idata, figsize=[4, 4])
plt.tight_layout()
plt.show()
az.plot_posterior(idata)
plt.show()