PCMCI#
PCMCI (PC algorithm + Momentary Conditional Independence) は、PC アルゴリズムを時系列用に拡張したもの。
高次元でもスケーラブル
非線形検定にも対応
目的#
多変量時系列 \(\{X_t^1, \ldots, X_t^N\}_{t=1}^T\) に対して、ラグ付き因果リンク \(X_{t-\tau}^j \to X_t^i\) つまり時系列 DAG(Time Series Graph, TSG)の推定がゴール。
アルゴリズム#
PCMCI は次の2段階で構成される。
PC₁ステップ(スクリーニング)
→ 候補親集合(potential parents)を削減する高速ステップMCIステップ(Momentary Conditional Independence)
→ 有意な因果リンクのみを確定する厳密な検定
PC₁ステップ:候補因果のスクリーニング#
PC-stableアルゴリズムを時系列データに適用したものを\(PC_1\)アルゴリズムという。このアルゴリズムにより原因(親ノード)の候補を探索する。
対象変数 \(X_{i,t}\) の候補親集合 \(\{ X_{j,t-\tau} \mid j = 1 \ldots N,\; \tau = 1 \ldots \tau_{\max} \}\) に対して独立性検定/条件付き独立性検定を行い、独立が疑われる候補リンクを削除していく。
ここでは「真の親集合を取りこぼさない」ことを重視するため、偽陽性のエッジも含む。
MCIステップ:因果リンクの最終判定#
PC₁で残った候補リンクに対して、より厳密な条件付き独立性検定を行い、偽陽性のエッジを削除する。
リンク \(X_{j,t-\tau} \to X_{i,t}\) を検定する
ここで
\(\text{pa}(X_{i,t})\), \(\text{pa}(X_{j, t-\tau})\) はそれぞれ \(X_{i,t}, X_{j, t-\tau}\) の親ノード
\(\text{pa}(X_{i,t}) \setminus \{X_{j,t-\tau}\}\) は \(X_{j,t-\tau}\) を除いた最大タイムラグ\(\tau\)までの\(X_{j,t}\)の親ノード
p-value < α → 因果リンクあり(採択)
point#
対象の親+候補親の親まで条件付けすることで交絡を減らす
スクリーニング済なので次元が低く、有意性検定のパワーが高い
PCMCI の利点#
1. 高次元でも高速
PC₁で候補が大幅に削減されるため、\(O(N^2 \tau_{\max})\) 程度の計算量で実用的。
高次元になりがちな気候データを扱った研究などでも使われている。
2. 強い自己相関・交絡への耐性
MCI の条件集合設計により、偽陽性(Type I error)を抑制。
発展モデル#
PCMCI+(拡張版)#
精度向上版で、追加機能:
同時刻(contemporaneous)因果も推定可能
v-structure の復元能力向上
非線形依存の検出性能がさらに改善
実装#
Tigramite パッケージで実行できる
from tigramite.data_processing import DataFrame
from tigramite.pcmci import PCMCI
from tigramite.independence_tests.parcorr import ParCorr
dataframe = DataFrame(data) # data: shape (T, N)
pcmci = PCMCI(
dataframe=dataframe,
cond_ind_test=ParCorr()
)
results = pcmci.run_pcmci(
tau_min=0, # 最小のラグを0(同時期の因果関係)
tau_max=5, # 最大のラグ
pc_alpha=0.05 # 独立性検定の有意水準
)
# 結果をplot
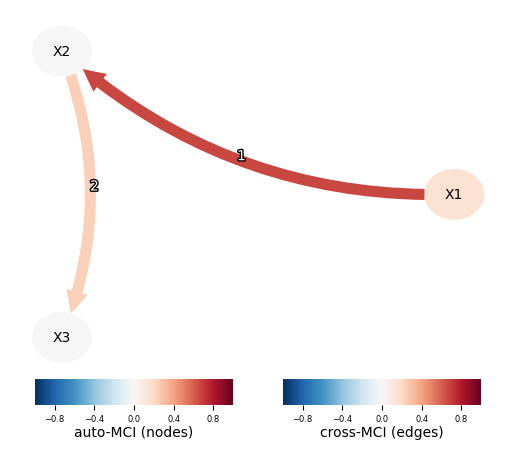
from tigramite import plotting as tp
tp.plot_graph(
val_matrix=results['val_matrix'],
graph=results['graph'],
var_names=["X1", "X2", "X3"],
link_colorbar_label='cross-MCI (edges)',
node_colorbar_label='auto-MCI (nodes)',
); plt.show()